Cross-kingdom RNA-Interferenz (RNAi) in der Pathogenbekämpfung
Bernhard Timo Werner
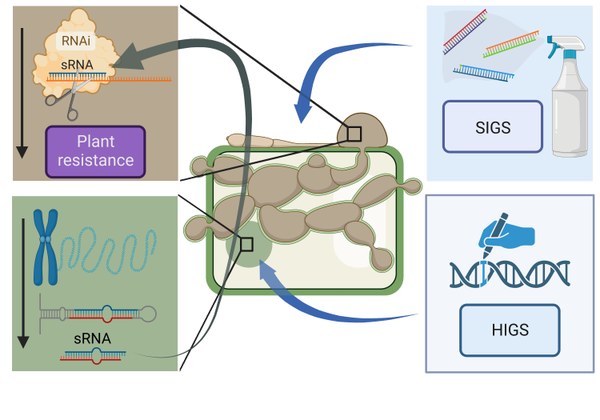
Hintergrund: Es ist bekannt, dass Pflanzen nicht-kodierende kleine regulatorische RNAs (sRNAs) mit antifungalen Eigenschaften produzieren. Diese sRNAs richten sich über den Mechanismus der cross kingdom RNAi (ckRNAi) gegen Pilzgene und führen zu einer Verringerung des Pathogenwachstums und der Krankheitsschwere. Die Gültigkeit dieses Konzepts wurde beispielsweise für den Pilzerreger Verticillium dahliae nachgewiesen, der bei mehr als 200 anderen Pflanzenarten verheerende Krankheiten mit Ertragsverlusten von bis zu 35 % verursacht. Darüber hinaus sind sRNAs wie Homologe der mikroRNA-159 aus Baumwolle konserviert und kommen in allen Landpflanzen vor. Sie hemmen das Pilzwachstum wirksam, indem sie das Isotrichodermin-C-15-Hydroxylase-Gen von V. dahliae zum Schweigen bringen. Während konstruierte sRNAs bereits routinemäßig in der Forschung und im kommerziellen Bereich zur Bekämpfung von Pflanzenkrankheiten eingesetzt werden, indem sie entweder über transgene Ansätze in der Pflanze selbst durch host-induced gene silencing (HIGS) exprimiert werden oder indem sRNAs auf Blätter aufgebracht werden (spray-induced gene silencing, SIGS), bleibt das Potenzial natürlich vorkommender sRNAs ungenutzt.
Projekt: Um diese ungenutzte Ressource zu nutzen, wollen wir sRNAs aus Weizen mit antifungalen Eigenschaften gegen die bekannten Pathogene Fusarium graminearum, Pyricularia oryzae und Puccinia striiformis identifizieren. Mit Hilfe der tiefen Sequenzierung des Transkriptoms, des Degradoms und des sRNA-Oms sollen ckRNAi-Ereignisse aufgeschlüsselt werden, um resistenzfördernde sRNAs zu finden. Funktionell validierte sRNAs werden Kandidaten für die Entwicklung einer neuen Generation induzierbarer künstlicher trans-acting small-interfering RNA (tasiRNA)-Vektoren mit einer breiten Resistenzaktivität gegen verschiedene Pilzpathogene sein. Die Nutzung natürlich vorkommender sRNAs im Pflanzenschutz birgt ein enormes Potenzial für die Entwicklung von Biokontrollmitteln, die dauerhaftere Resistenzen ohne unerwünschte Off-Target-Effekte auf die Umwelt verleihen.
Laborverfahren / Techniken: ckRNAi-seq, stem loop PCR, syn-tasiRNA, RLM-RACE, host-/spray-induced gene silencing.