Thema der Arbeitsgruppe
Innerhalb des gesamten Spektrums der am Menschen angewandten Arzneimittel unterrichten wir in der Lehre natürlich auch die komplexen Wirkmechanismen und die evidenzbasierte Anwendung aller heutzutage verwendeten antientzündlichen Medikamente.
Bei unseren Forschungsarbeiten fokussieren wir uns dagegen auf noch unverstandene Aspekte der Signaltransduktion von IL-1, TNF und anderen Stressstimuli. Hierzu zählen die Mechanismen der zytokin-induzierten Reprogrammierung der zellulären Genexpression, welche zur Aktivierung vieler weiterer Entzündungsgene führen und damit die Basis einer zellulären Entzündungsreaktion darstellen. Diese Prozesse spielen auch bei bakteriellen und viralen Infektion sowie Stressreaktionen eine wichtige Rolle, oft werden die Signalmoleküle hierbei redundant verwendet. Daher ermöglichen unsere Untersuchungen es IL-1 Systems auch eine sehr gute Vernetzung unserer Forschungsarbeiten mit den anderen Schwerpunkten der JLU wie dem Excellence Cluster Cardiopulmonary System (ECCPS) oder dem Universities of Giessen Marburg Lung Center (UGMLC).
Die Kontrolle der Genexpression erfordert ein komplexes Zusammenspiel von multiplen Signalwegen bei der Induktion und Termination der mRNA Synthese im Zellkern und des mRNA Abbaus im Zyoplasma. Wir studieren daher sowohl die nukleären Signalprozesse, die am Chromatin ablaufen als auch die Bildung von Protein:RNA Komplexen im Zytoplasma. Einen unserer Schwerpunkte bildet hierbei auch die Analyse reversibler posttranslationaler Modifikationen an Signalproteinen (Phosphorylierungen, Ubiquitinylierung, Acetylierungen), die als schnelle molekulare Schalter fungieren, um Funktionen von Proteinen bei der zellulären Entzündungsreaktion rasch an- oder abzuschalten. Diese Analysen erfordern Modellsysteme, die für die Isolation ausreichend großer Zellmengen geeignet sind. Daher arbeiten wir zurzeit schwerpunktmäßig in humanen oder murinen Zellkulturmodellen. Mit den in unserem Labor etablierten biochemischen, molekularbiologischen, zellbiologischen und bioinformatischen Methoden können wir vom Rezeptor bis zum Zellkern die Signalprozesse vermessen und häufig auch quantitativ erfassen. Mit modernen molekularbiologischen Methoden (RNAi, CRIPSR-cas) verändern wir dabei einzelne Schritte der Signaltransduktion und versuchen so, kausale Zusammenhänge und Mechanismen zu verstehen.
Im Folgenden ist eine schematische Darstellung der IL-1 Signaltransduktion in einer Zelle dargestellt. Die in die Abbildung integrierten Links demonstrieren Ergebnisse zur IL-1 Signaltransduktion, wie sie in unserem Labor erhoben werden.
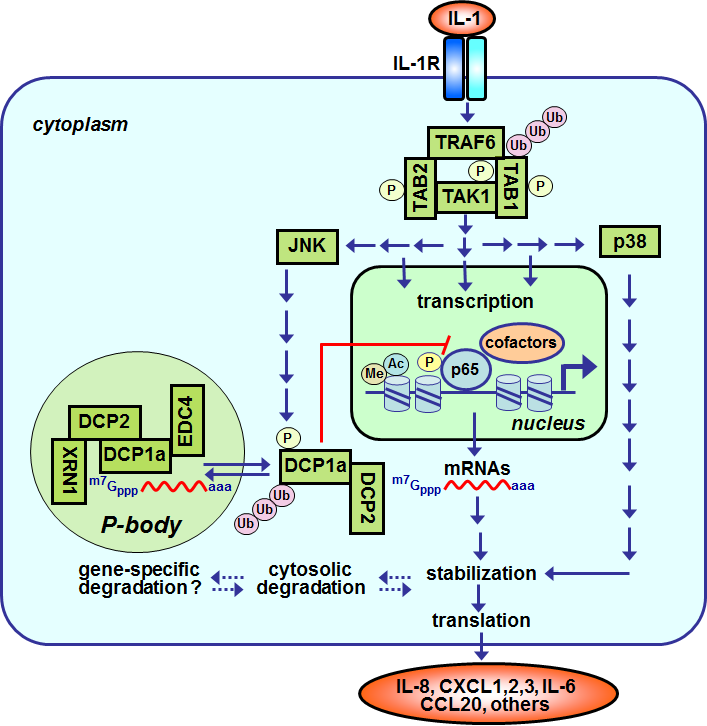
Schematische Darstellung der Aspekte der IL-1 Signaltransduktion, die wir zur Zeit bearbeiten
IL-1 Rezeptoren (IL-1R) werden als Heterodimere auf den meisten Körperzellen konstitutiv exprimiert. Dagegen werden die agonistischen Liganden (IL-1α und IL-1β) im Rahmen einer Entzündungsreaktion von Zellen des unspezifischen Immunsystems (z.B. Monozyten, Makrophagen) neu gebildet. Bindung von IL-1α oder IL-1β an den IL-1R induziert sodann über hier nicht gezeigte Protein:Protein Wechselwirkungen und rezeptorproximale Adaptorproteine und Proteinkinasen (MYD88, IRAK1-4, Tollip) innerhalb von 2-10 Minuten die Ausbildung eines oligomeren Signaltransduktionskomplexes, der aus dem Adaptorprotein TRAF6, der Proteinkinase TAK1 und den TAK1-interagierenden Proteinen TAB1-TAB3 besteht. TRAF6 hat hierbei Ubiquitinligaseaktivität, wobei die quervernetzten Ubiquitinketten Protein:Protein Wechselwirkungen mit weiteren Effektoren in der Signalkette stabilisieren (für ein Beispiel siehe hier). TAK1 und TAB1-TAB3 werden IL-1-abhängig reversibel phosphoryliert. Für ein experimentelles Beispiel der schnellen IL-1-abhängigen Signalübermittlung dieses Signalkomplexes siehe hier (Aktivierung von TAK1). Mit zeitlicher Verzögerung werden sodann innerhalb von 10-30 Minuten die MAP Kinasen JNK, p38 und ERK (hier nicht gezeigt) und der NF-κB Signalweg aktiviert (Aktivierung von p65 (1), (2)). JNK phosphoryliert und aktiviert Transkriptionsfaktoren aus der AP-1 Familie wie z.B. c-Jun (Aktivierung von JNK). Zellen, denen JNK oder c-Jun fehlen, zeigen starke Defekte in der Regulation IL-1-abhängiger Gene siehe hier. NF-κB ist ein komplex regulierter Transkriptionsfaktor, der in seiner inaktiven Form im Cytosol verankert ist und nach IL-1 Aktivierung in den Zellkern transloziert (Link Abb 5.A), um an spezifische NF-κB Bindungsstellen in den regulatorischen Bereichen (Promotoren und Enhancern) von vielen entzündungsrelevanten Genen zu binden. Für ein Beispiel der IL-1-abhängigen NF-κB Bindung an den IL8 Promoter siehe hier. Für eine Übersichtsabbildung der IL-8 Generegulation siehe hier. 2002.pdf). Für unsere Ansätze, bei denen wir genomweite Effekte von IL-1 untersuchen, siehe die Darstellung eines Mikroarray Experimentes. Für ein Beispiel der genomweiten Untersuchungen von Protein:DNA Komplexen mittles ChIP-seq siehe hier. Zusätzlich zur Gentranskription reguliert IL-1 die Bildung von zytoplasmatischen processing-bodies, welche aus Protein:RNA Komplexen aufgebaut sind, u.a. Decapping Faktoren enthalten und am Abbau von mRNAs beteiligt sind (für ein Beispiel siehe hier). Der bisher bekannte Teil des IL-1 Signalsystemes enthält mehr als 100 Komponenten, die wir zu einem Netzwerk verbunden und mit mathematischen Operatoren verbunden haben, um den Signalfluss besser modellieren zu können. Dieses Signalsystem zeigt außerdem vielfachen Crosstalk zu anderen Signalsystemen, wie z.B. dem Zellzyklus (siehe IL-1, Zellzyklus und CDK6).
Abkürzungen: IL-1R, interleukin-1 receptor; TRAF6, TNF-receptor-associated factor 6; TAK1, TGFβ-activated protein kinase 1; TAB1/2, TAK1-binding protein1/2; JNK, Jun-N-terminal kinase; NF-κB, nuclear factor kappa B; Pol II, RNA polymerase II; P, phosphorylation; ac, acetylation; ub, ubiquitination; XRN1, 5'-3' Exoribonuclease 1; DCP1a, decapping enzyme homolog 1 a; DCP2, decapping enzyme homolog 2; EDC4, enhancer of mRNA decapping 4.
