Projekt 1: Erstellung eines Zellatlas des Leberegels durch Einzelzell-RNA-Sequenzierung
Klassische Transkriptom Methoden mangelt es an zellulärer Auflösung
Mittels RNA Sequenzierung (RNAseq) lässt sich die Transkription aller Genen eines Organismus messen. Klassischen Ansätzen verwenden für diese Methoden komplexe Gewebeproben die aus unterschiedlichen Zellen zusammmengesetzt sind. Durch den Aufschluss des Gewebes entsteht ein Homogenat der Zellen und die gemessene Genexpression stellt daher einen Mittelwert der Transkription eines Gens dar. Innovative Methoden wie “single-cell RNAseq” (sc-RNAseq) erfassen Transkripte in einzelnen Zellen. Der so generierte Datensatz besticht durch höhere Auflösung und erlaubt genauere Messung von Genaktivität in einzelnen Zelltypen.
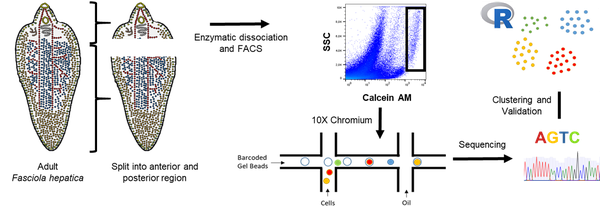
Ein Zell Atlas für den Leberegel Fasciola hepatica
In diesem Projekt arbeiten wir an der Kartierung von Zelltypen basierend auf ihrer Transkription. Die ermöglicht uns die funktionelle Analyse dieser Zellen und einem tieferen Verständnis der Biologie dieser Parasiten.
Diese innovative Methode bietet neben spannenden Applikationen in der medizinischen Forschung auch eine einzigartige Möglichkeit für die Forschung an neuen Organismen. Während für klassische Modellorganismen wie zum Beispiel Maus, Fruchtfliege oder der Fadenwurm Caenorhabditis elegans seit Jahrzehnten im Zentrum der Forschung standen und Daten zu verschiedenen Zelltypen verfügbar sind, ist dies für viele anderen Organismen nicht der Fall. Hochdurchsatz Methoden wie sc-RNAseq bieten die Möglichkeit eine große Menge an Information zu bekommen.

Bild von dissozierten Zellen des Leberegels in A: Hellfeldmikroskopie und B: Fluoreszenmikroskopie
mit Farbstoff Calcein
